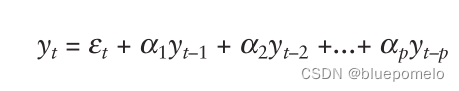微生物交互关系研究论文摘要集锦
- 296. A computational exploration of bacterial metabolic diversity identifying metabolic interactions and growth-efficient strain communities
- 297. A Modified Entropy-Based Approach for Identifying Gene-Gene Interactions in Case-Control Study
- 298. A new mathematical model of bacterial interactions in two-species oral biofilms
- 299. A Novel Evolution-Based Method for Detecting Gene-Gene Interactions
- 300. Active learning for human protein-protein interaction prediction
- 115. @MInter: automated text-mining of microbial interactions.
- 301. Characterizing metabolic interactions in a clostridial co-culture for consolidated bioprocessing
- 302. Competition, Not Cooperation, Dominates Interactions among Culturable Microbial Species
- 7. Competitive and cooperative metabolic interactions in bacterial communities
- 65. Complex Species Interactions and the Dynamics of Ecological Systems: Long-Term Experiments
- 303. CompNet: a GUI based tool for comparison of multiple biological interaction networks
- 304. Computing paths and cycles in biological interaction graphs
- 305. Detecting temporal protein complexes from dynamic protein-protein interaction networks
- 306. Dynamical modeling of uncertain interaction-based genomic networks
- 307. Effective comparative analysis of protein-protein interaction networks by measuring the steady-state network flow using a Markov model
- 24. Elucidation of complexity and prediction of interactions in microbial communities
- 173. Establishment and metabolic analysis of a model microbial community for understanding trophic and electron accepting interactions of subsurface anaerobic environments
- 308. Exploring biological interaction networks with tailored weighted quasi-bicliques
- 309. Genome-based Modeling and Design of Metabolic Interactions in Microbial Communities
- 310. Global metabolic interaction network of the human gut microbiota for context-specific community-scale analysis
- 311. Host-Gut Microbiota Metabolic Interactions
- 312. Host-microbial interaction in the mammalian intestine and their metabolic role inside
- 313. Host-microbial interactions in the metabolism of therapeutic and diet-derived xenobiotics
- 314. Identification of novel metabolic interactions controlling carbon flux from xylose to ethanol in natural and recombinant yeasts
- 315. Inference of Network Dynamics and Metabolic Interactions in the Gut Microbiome
- 316. Inferring microbial interaction network from microbiome data using RMN algorithm
- 317. Inferring microbial interaction networks from metagenomic data using SgLV-EKF algorithm
- 318. Interaction networks for identifying coupled molecular processes in microbial communities
- 319. Large-scale in silico modeling of metabolic interactions between cell types in the human brain
- 320. Limitations of a metabolic network-based reverse ecology method for inferring host–pathogen interactions
- 321. Metabolic Interaction of Helicobacter pylori Infection and Gut Microbiota
- 322. Metabolic interactions in microbial communities: untangling the Gordian knot
- 323. Metabolic network analysis reveals microbial community interactions in anammox granules
- 324. MetaMIS: a metagenomic microbial interaction simulator based on microbial community profiles
- 325. Microbial interactions for enhancement of a-amylase production by Bacillus amyloliquefaciens 04BBA15 and Lactobacillus fermentum 04BBA19
- 326. Microbial interactions: from networks to models
- 327. MIMO: an efficient tool for molecular interaction maps overlap
- 328. MMinte: an application for predicting metabolic interactions among the microbial species in a community
- 48. Model-based quantification of metabolic interactions from dynamic microbial-community data
- 76. Multispecies communities: interspecies interactions influence growth on saliva as sole nutritional source.
- 329. PID: the Pathway Interaction Database
- 16. Predicting microbial interactions through computational approaches
- 330. The integrated analysis of metabolic and protein interaction networks reveals novel molecular organizing principles
- 14. The large-scale organization of the bacterial network of ecological co-occurrence interactions
- 331. The linear neighborhood propagation method for predicting long non-coding RNA –protein interactions
- 332. Understanding the interactions between bacteria in the human gut through metabolic modeling
- 333. Visualization of Metabolic Interaction Networks in Microbial Communities Using VisANT 5.0
- 364. Interaction networks, ecological stability, and collective antibiotic tolerance in polymicrobial infections
- 366. Lotka-Volterra pairwise modeling fails to capture diverse pairwise microbial interactions
296. A computational exploration of bacterial metabolic diversity identifying metabolic interactions and growth-efficient strain communities
296.细菌代谢多样性识别代谢相互作用和高效生长菌株群落的计算性探索
摘要:背景:代谢相互作用涉及代谢产物在微生物物种之间的交换。大多数微生物生活在社区,通常依靠代谢相互作用来增加养分供应,更好地利用特定的环境。基于约束的模型已经成功地分析了细胞代谢并描述了基因型-表型关系。但是,对基因组规模的多种群相互作用的研究还很少。基于基因组规模的方法,我们提出一个图形理论的方法与代谢模型,以探讨细菌菌株之间的代谢变异性,并鉴定和描述代谢相互作用的菌株群落由两个或两个以上菌株组成的分批培养。我们证明了我们的方法在不同的单碳源条件下对细菌大肠杆菌的适用性。结果:为每个生长条件构建不同的多样性图。所构建的图的图论性质反映了细胞固有的高代谢冗余性以单基因敲除,揭示了关于副产物的独特代谢能力的突变体集合体,在各种条件下展示了一致的代谢性行为并且显示出朝向建立多态性,同时表明由特定适应特定条件的菌株组成的群落更有可能进化。我们揭示了相对于相应的单一栽培而言,即使菌株群落没有被建模成为集体目标(例如群落生长)的运作,我们也发现了相对于相应的单一栽培而言生长得到改善的菌株群落,并且我们确定了在这些批次共培养物中交换的代谢物的范围。结论:本研究提供了大肠杆菌菌株在不同条件下副产物代谢变异的全基因组描述,并展示了如何利用代谢差异来鉴定代谢相互作用的菌株群落。这项工作还扩展了现有的化学计量学模型,以描述批次共培养,并提供了一个菌株群体的代谢相互作用的程度,揭示其对增长的重要性。
297. A Modified Entropy-Based Approach for Identifying Gene-Gene Interactions in Case-Control Study
297.一个识别基因-基因相互作用的基于修正熵的方法的病例对照研究
摘要:基因 - 基因相互作用可能在复杂疾病的遗传学中起重要作用。基因 - 基因相互作用的检测和表征是一个具有挑战性的问题,激发了各种统计方法的发展来解决它。在这项研究中,我们介绍一种方法来测量基因相互作用使用基于熵的统计从特质和基因型组合的列联表。我们还使用图形开发了一个勘探程序。我们提出了一个标准化的相对信息增益(RIG)测量来评估单核苷酸多态性(SNP)组合之间的相互作用。为了识别第k阶相互作用,构建了k个SNP的性状和基因型组合的列联表,其中计算了RIG。使用置换数据集的均值和标准差对RIG进行标准化。选择产生高标准RIG的SNP组合用于基因 - 基因相互作用。通过使用标准化的RIG,可以检测高阶交互作用和不同阶次之间交互强度的比较。我们已经将所提出的标准化的基于熵的方法应用于来自模拟研究和真正的遗传关联研究的两种类型的数据集。我们通过对不同外显率,SNP数量和样本大小的不同遗传模型的功效分析,比较了我们的方法和多因素降维法(MDR)。我们的方法显示在模拟和真实遗传数据中成功鉴定遗传关联和基因 - 基因相互作用。仿真结果表明,所提出的基于熵的方法能够更好地检测高阶相互作用,并且在大多数情况下优于MDR方法。所提出的方法非常适合于检测没有主效应的交互以及包括主效应的模型。
298. A new mathematical model of bacterial interactions in two-species oral biofilms
298.两种口腔生物膜中细菌相互作用的一种新的数学模型
摘要:牙周炎是细菌性炎性疾病,其中存在于牙齿支持组织上的细菌生物膜从健康状态转变为致病状态。在涉及该疾病的细菌物种中,已经显示牙龈卟啉单胞菌(Porphyromonas gingivalis)诱导生态失调,并诱导健康细菌如格氏链球菌(Streptococcus gordonii)的毒力。在生物膜发育过程中,主要的定居者如格氏链球菌(S.gordonii)首先附着在表面上,并随后粘附牙周病原体如牙龈卟啉单胞菌(P.gingivalis)。这两种细菌之间的相互作用已经在生物膜的粘附步骤中被广泛研究。该研究的目的是使用数学模型来理解在生物膜的生长阶段期间两种物质的相互作用,其中对于这些生物膜几乎没有可用的知识。这种两物种生物膜模型是基于依赖于底物的生长,实施损伤参数,并验证了实验生物膜获得的数据。提出了三种不同的相互作用假设,并使用该模型进行检验:独立性,两种细菌种类之间的竞争,或者一个物种对其他物种的毒性的诱导。在最后的假设数学模型中发现了实验模拟生物膜和模拟生物膜之间的充分性。两种细菌生物膜的这种新的数学模型依赖于不同的生长基质,可以应用于任何细菌种类,环境条件或生物膜发育的步骤。探索生物膜条件下的细菌相互作用将是非常有意义的。
299. A Novel Evolution-Based Method for Detecting Gene-Gene Interactions
299.一种检测基因-基因相互作用的新的基于进化的方法
摘要:背景:大规模SNP芯片技术的快速发展为我们阐明复杂疾病的遗传基础提供了巨大的机遇。大规模相互作用分析的方法已经从多个方面得到了发展。由于几个难题(例如,高维数据的稀疏性和低复制或验证率),开发用于检测各种形式的基因 - 基因相互作用的快速,强大和可靠的方法仍然是一个具有挑战性的任务。方法/主要发现:在这篇文章中,我们已经开发了一种基于进化的方法来搜索病例对照设计中的全基因组上位性上位。从进化的角度来看,我们认为人类疾病起源于古代的突变,并且认为潜在的遗传变异体在将人类分化为健康和患病方面发挥作用。基于这个概念,传统的进化测度,固定指标(Fst)的两个非连锁的基因座,测量人口之间的遗传距离,应该能够揭示疾病性状的负责任的遗传interplays。为了验证我们的建议,我们首先通过广泛的模拟研究了Fst的理论分布。然后,我们探索了通过SNP标记检测基因 - 基因相互作用的能力,并将其与传统的Pearson卡方检验,基于互信息的检验和基于连锁不平衡的几种疾病模型进行了比较。所提出的基于进化的方法在显性和加性模型中优于这些比较方法,不管疾病等位基因频率如何。然而,其隐性模式的表现相对较差。最后,我们将所提出的基于进化的方法应用于已发布数据集的分析。我们的研究结果表明,基于Fst的统计量的P值小于基于LD的统计量或泊松回归模型的P值。结论/意义:随着大规模遗传关联研究的快速发展,所提出的基于进化的方法可能成为鉴定上位性效应的有希望的工具。
300. Active learning for human protein-protein interaction prediction
300.主动学习以预测人类蛋白质-蛋白质相互作用
摘要:背景:细胞中的生物学过程是通过蛋白质 - 蛋白质相互作用进行的。确定一对蛋白质是否通过湿实验室实验相互作用是资源密集型的;今天只有约三万八千次互动,在几十万次预期的互动中出现。主动机器学习可以指导蛋白质对的选择以用于未来的实验表征,以加速对人类蛋白质相互作用组的准确预测。结果:随机森林(RF)以前被证明是有效的预测蛋白质相互作用。在这里,设计了四种不同的主动学习算法来选择用于训练RF的蛋白质对。使用这里描述的四种主动学习方法中的任何一种来选择少至500个蛋白质对的标签,分类器比3000个随机选择的蛋白质对获得更高的F分数(调和平均值和精确度)。与随机选择数据相比,主动学习的预测交互F分数显示增加约15%。结论:主动学习算法使学习更准确的分类器与更少的标签数据,并证明是有用的应用程序,其中手动注释的数据是强大的。这里展示的主动学习技术也可以应用于其他蛋白质组学应用,例如蛋白质结构预测和分类。
115. @MInter: automated text-mining of microbial interactions.
115. @Minter:微生物相互作用的自动文本挖掘
摘要:动机:微生物联盟经常被社区内的众多互动所定义,这是理解其功能的关键。虽然微生物相互作用已被广泛研究实验,关于他们的信息分散在科学文献。由于手工整理是不可行的选择,因此需要使用自动数据处理工具来使这些信息易于访问。结果:我们提供了@MInter,一个基于支持向量机的自动化信息提取系统,用于分析论文摘要和推断微生物的相互作用。 @MInter在735种物种相互作用的手工策划的金标准数据集上被训练和测试,并且被构造作为这项研究的一部分3917注释的摘要。交叉验证分析显示@MInter能够检测出与一种或多种微生物相互作用有关的摘要,特异性高(特异性= 95%,AUC = 0.97)。尽管在抽象中识别特定微生物相互作用方面存在挑战(相互作用水平回忆= 95%,精度= 25%),@MInter显示与其他方法相比,annotator工作量减少了13倍。将@MInter应用于人类皮肤上丰富的175种细菌,我们确定了357篇文献报道的微生物相互作用的网络,证明了其用于研究微生物群落的效用。
301. Characterizing metabolic interactions in a clostridial co-culture for consolidated bioprocessing
301.表征梭菌共培养中的代谢相互作用以强化生物过程
摘要:含有纤维素分解和溶剂性物质的梭菌共培养是用于从纤维素生物质生产生物化学物质和生物燃料的潜在联合生物处理(CBP)方法。已经证实,与解纤维素梭菌的单一培养物(BL 5:119-124,1983)相比,丙酮丁醇梭菌和解纤维素梭菌的共培养物中的纤维素利用率得到改善。然而,这种共培养中的代谢相互作用尚不清楚。为了研究这种共培养中的代谢相互作用,我们使用qPCR动态地表征生理学和微生物组成。qPCR数据显示与单一培养物相比,共培养物中解纤维素酵母的生长速率更高。我们的结果还表明,与在与共培养的条件相似的条件下不显示任何纤维素分解活性的解纤维素梭菌的单一培养物相反,共培养确实显示出纤维素分解活性甚至优于解纤维素梭菌单在7.2的最佳pH下培养。此外,实验表明共培养纤维素分解活性取决于共培养物中丙酮丁醇梭菌的浓度,因为在低浓度的丙酮丁醇梭菌中没有观察到纤维素分解活性,从而证实了丙酮丁醇梭菌在改善共培养物中的解纤维素梭菌的生长。此外,在共培养批次实验中检测到350mg / L的丁醇浓度。这些结果表明这两个物种之间存在协同作用,而丙酮丁醇梭菌的代谢活性显着提高了共培养物中的纤维素分解活性,并且允许解纤维素梭菌在恶劣的共培养条件下存活,其不允许解纤维素梭菌独立地生长和代谢纤维素。丙酮丁醇梭菌很可能通过代谢产物如丙酮酸的交换而改善共培养中的解纤维素梭菌的纤维素分解活性,使其能够在苛刻的共培养条件下生长和代谢纤维素。
302. Competition, Not Cooperation, Dominates Interactions among Culturable Microbial Species
302.竞争,而不是合作,主导可培养微生物物种的相互作用
摘要:总结:微生物细胞分泌大量的酶,清除分子和促进其他细胞生长和生存的信号[1-4]。这种观察与物种间合作的进化是一致的[5],现在越来越强调不同微生物之间合作的重要性[4,6]。然而,我们缺乏对不同物种之间相互积极互动的重要性的系统测试,这对于评估自然社区合作进化的共性和重要性至关重要。在这里,我们研究了从一个普通的水生环境中分离出来的细菌菌株之间的相互作用的程度。利用从两个独立实验收集的数据评估多样性梯度上的群落生产力,我们表明:(1)在成对物种组合中,绝大多数的相互作用是净负的;(2)没有证据表明强时高阶正效应出现两种以上的物种混合在一起。我们的数据并不排除在一个物种获得另一个物种而牺牲另一个物种的方向上产生积极影响的可能性,即捕食类似猎物的相互作用。然而,这些不构成合作,我们的分析表明,适应其他微生物物种的典型结果将是竞争性的,而不是合作的表型。
7. Competitive and cooperative metabolic interactions in bacterial communities
7.细菌群落中竞争和协作代谢相互作用
摘要:揭示塑造社区的生态原则是后基因组时代的一大挑战。迄今为止,缺乏描述物种间相互作用的系统方法。在这里,我们独立预测了来自118个物种代谢模型集合的6,903个细菌对之间的竞争和合作潜力。我们绘制了竞争与合作之间错综复杂的联系,表明在适度的资源重叠程度下合作潜力是最大化的。利用2,801个样本的生态数据,我们探讨了细菌相互作用与共存模式之间的关系。在互相排斥的物种间观察到的高度竞争支持竞争在社区组装中的作用。合作互动通常是单向的,对提供者没有明显的益处。然而,在自然界中,细菌通常会形成紧密的合作循环,从而对涉及的所有物种间接获益。这些发现对于将来的生物修复和生物生产应用优化的团簇设计非常重要。
65. Complex Species Interactions and the Dynamics of Ecological Systems: Long-Term Experiments
65.复杂物种相互作用和生态系统动力学:长期实验
摘要:将实验操作与长期数据收集相结合的研究揭示了影响生态系统结构和动态的物种之间的精细相互作用。美国沙漠灌木林和松嫩桧柏林的研究表明,(1)物种种群的复杂动态反映了与其他生物的相互作用和气候波动; (ii)基因型x环境相互作用影响物种对环境变化的反应; (3)主要植物物种的草食动物抗性特征,以及通过该系统梯级动物物种的影响,以影响许多生物和生态系统过程; (4)一些环境扰动可能导致生态系统的大规模重组,因为它们超过了主要或重要物种的生态容忍度,而其他变化则可能由于补充物种的补偿动态而被缓冲。
303. CompNet: a GUI based tool for comparison of multiple biological interaction networks
303.CompNet:一个用于比较多种生物相互作用网络的GUI工具
摘要:背景:网络可视化和分析工具有助于更好地理解复杂的生物系统。此外,为了理解各种环境条件(例如压力,感染)下系统的行为差异,需要比较多个网络。多重网络之间的这种比较可能有助于主张因果关系并确定所研究的生物系统的关键组成部分。尽管存在许多可用的网络比较方法,它们使用诸如网络对齐和查询之类的技术来计算所选网络之间的成对相似性,但是它们中的大多数在多个网络的交互式视觉比较方面具有有限的特征。结果:在本文中,我们介绍CompNet - 一个基于图形用户界面的网络比较工具,它允许基于各种网络度量的多个网络的可视比较。 CompNet允许选定的一组网络的联合,相交和/或互补区域的交互式可视化。不同的可视化特征(例如,馅饼节点,边缘馅饼矩阵等)有助于容易地识别在所比较的网络中的关键节点/交互及其意义。该工具还允许在组成节点和社区组成的邻域结构的基础上进行网络比较,这是分析生物网络时特别有用的特征。为了证明CompNet的效用,我们比较了一个(时间序列)人类基因表达数据集,感染两种结核分枝杆菌后,重叠在人类蛋白质 - 蛋白质相互作用网络上。使用CompNet的各种功能不仅使我们能够理解感染过程中相互作用模式的变化,还有助于推断两种菌株感染宿主细胞的可能命运。结论:CompNet有望成为一种有价值的可视化数据挖掘工具,可免费从http://metagenomics.atc.tcs.com/compnet/或http://121.241.184.233/compnet/
304. Computing paths and cycles in biological interaction graphs
304.计算生物相互作用图中的路径和循环
摘要:背景:交互图(带符号的有向图)为系统生物学提供了重要的定性建模方法。它们可以分析细胞网络中的因果关系,甚至可以用来预测系统动力学的定性方面。交互图分析的基本问题是枚举路径和周期(反馈回路)以及计算最短的正/负路径。这些计算问题在系统生物学的范围内只有很小程度的讨论,特别是最短的符号路径问题需要算法的发展。结果:我们首先回顾了用于枚举路径和循环的算法,并且显示这些算法优于最近提出的基于基本模式计算的列举方法。这项工作的主要部分处理最短的正/负路径的计算,一个NP完全问题,其中只有很少的算法在文献中描述。我们提出扩展和几个新的算法变种来计算精确结果或近似值。与各种具体的生物网络的基准表明,有时可以在具有几百个节点的网络中获得准确的结果。一类更大的图仍然可以通过结合穷举和简单搜索策略的新算法精确处理。对于精确解的计算变得耗时或不可行的图,我们设计了一个具有多项式复杂度的近似算法。引人注目的是,在现实网络(可以与精确结果进行比较的情况下),该算法提供了非常接近或等于确切值的结果。这种现象可能是由于包含相对较少数量的负反馈回路的蜂窝信令和调节网络的特定拓扑结构所致。结论:交互图中最短正负路径和周期的计算是系统生物学网络分析的重要手段。这个贡献吸引了社区对这个重要的计算问题的关注,并提供了一些新的算法,部分专门针对生物相互作用图。所有算法都已经在CellNetAnalyzer框架中实现,可以在http://www.mpi-magdeburg.mpg.de/projects/cna/cna.html下载用于学术用途。
305. Detecting temporal protein complexes from dynamic protein-protein interaction networks
305.从动态蛋白质-蛋白质相互作用网络中检测临时蛋白质复合物
摘要:背景:蛋白质动态相互作用,以执行其生物学功能。蛋白质相互作用网络(PPI)的动态操作也反映在蛋白质复合物的动态形成中。现有的蛋白质复合物检测算法通常忽略了PPI网络中蛋白质相互作用的固有时间性质。系统分析颞叶蛋白复合物,不仅可以提高蛋白质复合物检测的准确性,而且可以加强我们对细胞组织动态蛋白组装过程的生物学知识。结果:在这项研究中,我们提出了一种新的计算方法来预测时间蛋白质复合物。具体而言,我们首先通过时间基因表达数据和蛋白质相互作用数据的联合分析来构建一系列动态PPI网络。然后提出了一个时间平滑重叠复合检测模型(TS-OCD),用于从这些动态PPI网络中检测时间蛋白质复合物。TS-OCD自然可以捕获连续时间点之间网络的平滑性,并在每个时间点检测重叠的蛋白质复合物。最后,引入了一个基于非负矩阵分解的算法,将不同时间点的这些非常相似的时态复合体进行合并。结论:广泛的实验结果表明,提出的方法是非常有效的检测时间蛋白复合物比最先进的复杂检测技术。
306. Dynamical modeling of uncertain interaction-based genomic networks
306.基于不确定相互作用的基因组网络的动态建模
摘要:背景:基因组网络的大多数动力学模型是基于两种现有的方法建立的,一种基于过程,另一种基于布尔型网络。当涉及实验室的实验设计目的时,两者都是有问题的。第一种方法需要全面了解涉及所有生物学过程的参数,而第二种方法的结果可能没有生物学上的一致性,因此不能在实验室中进行测试。而且,目前的方法不能很好地利用现有的策划知识库,不会考虑知识的不确定性。因此,需要一种新的方法来生成一个基于现有生物数据的动力学模型,假设不确定性,而实验设计的结果可以在实验室中进行检验。结果:我们提出了一种新的基因组网络动态建模方法,可以利用公共数据库提供的交互知识。该模型为物理实体分配离散状态,基于数据库中提供的信息在交互之间设置优先级,并基于关联的节点状态更新每个交互。只要动态出现不确定性,就会探索所有可能的结果。通过使用所提出的模型,生物学家可以研究对于手动分析来说太复杂的调节网络。结论:所提出的方法可以有效地用于构建基于交互的基因组网络的动力学模型,而不需要对影响网络动力学的所有参数有完整的了解,因此基于一小部分可用数据。
307. Effective comparative analysis of protein-protein interaction networks by measuring the steady-state network flow using a Markov model
307.通过使用马尔科夫模型测量稳态网络流来有效比较蛋白质-蛋白质相互作用网络
摘要:背景:蛋白质 - 蛋白质相互作用(PPI)网络的比较分析提供了检测跨不同物种的保守功能网络模块的有效手段。这样的模块通常由具有保守相互作用的直向同源蛋白组成,可以利用这些蛋白通过网络比较来计算预测模块。结果:在这项工作中,我们提出了一个新的概率框架比较PPI网络,并有效地预测蛋白质之间的对应关系,表示为网络节点,属于给定PPI网络的保守功能模块。其基本思想是基于马尔可夫随机游走模型来估计属于不同PPI网络的节点之间的稳态网络流量。随机游走者被设计为随机移动到PPI网络内的相邻节点以及具有高序列相似性的潜在直系同源节点之间的跨网移动。基于这个马尔科夫随机游走模型,我们估计稳态网络流量或者随机游走者所做的转换的长期相对频率 - 在不同的PPI网络中的节点之间,这可以用作测量它们的概率分数潜在的对应。随后,估计的分数可以用于通过网络比对来检测保守功能模块中的直系同源蛋白。结论:通过基于多个实际PPI网络的评估,我们证明了所提出的方案导致改进的比对结果在生物学上更有意义,并且计算成本降低,优于当前的最新算法。源代码和数据集可以从http://www.ece.tamu.edu/~bjyoon/CUFID下载。
24. Elucidation of complexity and prediction of interactions in microbial communities
24.阐明微生物群落的复杂性和相互作用的预测
摘要:微生物与微生物群落的其他成员,高等生物以及它们的环境发生复杂的相互作用。然而,确定这些相互作用的确切性质可以是具有挑战性由于大量在这些社区的成员的和相互作用的歧管就可以搞英寸的各种组学的数据,如16S rRNA基因测序,猎枪宏基因组学,宏转录组学,宏蛋白质组学和代谢组学已被用于解释社区结构,相互作用和由此导致的原位社区动态。对这些多元数据的解释通常需要先进的计算方法。建模方法是整合,上下文化和解释实验数据的强大工具,从而揭示了微生物组成的基本过程。在这里,我们审查目前的方法和方法,包括实验和计算,澄清微生物群落中的相互作用,并预测其对扰动的反应。
173. Establishment and metabolic analysis of a model microbial community for understanding trophic and electron accepting interactions of subsurface anaerobic environments
173理解亚表层厌氧环境下营养和电子接受相互作用的模型微生物群落的建立和代谢分析
摘要:微生物群落控制着重要的生物地球化学循环的速率,对于生物技术,生物修复和工业微生物过程是重要的。出于这个原因,我们构建了一个模型微生物群落,由三个依赖于营养相互作用的物种组成。三种微生物群落由解纤维素梭菌(Clostridium cellulolyticum),Desulfovibrio vulgaris Hildenborough和Geobacter sulfurreducens组成,并在连续培养条件下生长。纤维二糖作为解纤维素酵母的碳源和能源来源,而D. vulgaris和G. sulfurreducens从纤维二糖发酵的代谢产物中获得碳和能量,分别提供硫酸盐和富马酸盐作为电子受体。对培养物进行qPCR监测显示解纤维素酵母(C.cellulolyticum)如预期的那样具有显性,并证实了D.vulgaris和G.soreducens的存在。提出的三物种群落的碳和电子流动的代谢建模表明,解纤维梭菌和D. vulgaris的生长是电子供体有限的,而G. sulfurreducens是受电子受限的。结果表明解纤维梭菌(C. cellulolyticum),D. vulgaris和G. sulfurreducens可以在连续培养系统中的共培养中生长,其中D. vulgaris和G. sulfurreducens依赖于解纤维素梭菌(C. cellulolyticum)的代谢副产物的营养物。这代表着朝着开发由代表营养网络功能组的成员组成的易处理模式生态系统迈出的一步。
308. Exploring biological interaction networks with tailored weighted quasi-bicliques
308.用特制的加权准bicliques探索生物相互作用网络
摘要:背景:生物网络为基因及其产物的功能表征,DNA-蛋白质相互作用的表征,调节机制的鉴定以及其他生物学任务提供了基本的见解。由于实验和生物的复杂性,它们的计算开发面临着许多算法上的挑战。结果:我们引入了新颖的加权准biclique问题来识别生物网络中的功能模块,用二分图表示。与以前的准二元问题不同,我们通过使用边加权准二元曲来包含生物相互作用水平。虽然我们证明我们的问题是NP难题,但我们也描述了IP配方来计算中等大小网络的精确解。结论:我们使用模拟和经验数据来验证我们的IP解决方案的有效性。仿真结果表明,该算法具有较高的拟二次召回率,经验数据证实了加权准二元曲线提取特征和恢复生物网络缺失交互的能力。
309. Genome-based Modeling and Design of Metabolic Interactions in Microbial Communities
309.基于基因组的微生物群落建模和代谢相互作用设计
摘要:传统上,生物技术研究集中在被认为具有必要代谢功能的个体微生物菌株,或者具有引入这些功能的能力以实现特定任务。对于许多重要的应用来说,开发这种万能微生物是一项非常具有挑战性的工作,如果不是不可能的话。相比之下,大自然采用基于不同微生物物种的协同组合的完全不同的策略,这些组合共同实现期望的任务。这些自然社区已经发展到利用每个物种的天然代谢能力,并且对其环境中的变化高度适应。然而,由于缺乏合适的实验和计算工具,微生物群落被证明难以研究。
随着基因组测序,组学技术,生物信息学和基因组模型的出现,研究人员现在具有前所未有的分析和设计微生物群落代谢的能力。这次审查的目的是总结基因组规模代谢建模的最新应用微生物群落。对集总式社区模型的简要介绍用于激发个体物种及其代谢相互作用的基因组水平描述的需要。基因组尺度模型的回顾始于静态建模方法,适用于细胞外环境可以假定为时间不变或缓慢变化的社区。描述了静态建模方法的动态扩展,然后综述了用于设计合成微生物群落的基因组尺度模型的应用。综述总结了用于分析社区新陈代谢的宏基因组学工具以及未来研究的展望。
310. Global metabolic interaction network of the human gut microbiota for context-specific community-scale analysis
310.特定背景下群落尺度分析人类肠道微生物的全局代谢相互作用网络
摘要:复杂的微生物 - 微生物和宿主 - 微生物化学交叉的系统级框架将有助于阐明肠道微生物在健康和疾病中的作用。在这里,我们报告了一个文献策划的人类肠道微生物群的种间网络,称为NJS16。这是一个广泛的数据资源,由1/4570个微生物种类和3个人类细胞类型通过> 4400个小分子运输和大分子降解事件代谢相互作用组成。根据我们网络的内容,我们开发了一个数学方法来阐明给定人群(如疾病队列)肠道微生物群落的代表性微生物和代谢特征。将这一策略应用于来自2型糖尿病患者的微生物组数据揭示了肠道微生物生态系统的具体背景基础设施,具有较大代谢影响的核心微生物实体以及可能指示相关的社区代谢过程的经常产生的代谢化合物。我们的网络为人类肠道内社区规模微生物活动的综合调查奠定了基础。
311. Host-Gut Microbiota Metabolic Interactions
311.宿主肠微生物代谢相互作用
摘要:肠道微生物的组成和活性与来自出生的宿主一起发展,并且受到宿主基因组,营养和生活方式的复杂相互作用的影响。肠道微生物群参与多个宿主代谢途径的调节,产生互动的宿主 - 微生物代谢,信号传导和免疫炎症轴,生理上连接肠,肝,肌肉和大脑。对这些轴的更深入的理解是优化治疗策略来操纵肠道微生物群以对抗疾病和改善健康的先决条件。
312. Host-microbial interaction in the mammalian intestine and their metabolic role inside
312.哺乳动物肠内宿主-微生物相互作用及其代谢作用
摘要:哺乳动物的肠道已经与数百万年的微生物联合起来,这些微生物与我们的身体在身体上密切相关。从出生开始,这个人群就与肠道组织持续地密切接触。最近的研究结果表明,土着细菌在出生后早期的肠道发育中起着重要的诱导作用。这些发现揭示了哺乳动物肠道与其原核伴侣的相互作用,这对其正常发育是必不可少的。在它们的共同进化过程中,细菌微生物群已经建立了多种机制来影响真核宿主,通常以有利的方式,并保持其稳定的生态位。人类微生物群的原核生物基因组编码超出宿主基因组的一系列代谢能力,使得微生物群成为人类生理学的一个组成部分。在正常的肠胃微生物相互作用中获得对双方伙伴的更全面的了解,可能揭示这种关系如何发生偏差,并导致一系列免疫,炎症和代谢紊乱,并可能揭示这种关系可能被用于治疗目的的机制。这篇综述提供了这个令人兴奋的新兴领域的简要概述。
313. Host-microbial interactions in the metabolism of therapeutic and diet-derived xenobiotics
313.在治疗和饮食来源的宾主共栖生物代谢作用的宿主-微生物相互作用
摘要:我们相关的微生物群落在人类健康和疾病易感性中起着关键作用,但是它们也构成治疗干预的程度尚不清楚。在这里,我们整合了肠道微生物对治疗药物和饮食衍生生物活性化合物代谢的直接和间接影响的经典和当前研究的结果。我们特别注意微生物对宿主对异生物质反应的影响,并且越来越多的人认为治疗结果反映了我们与微生物世界的亲密伙伴关系,并提供了一个初步框架,从中考虑更全面的药理学和营养观点。
314. Identification of novel metabolic interactions controlling carbon flux from xylose to ethanol in natural and recombinant yeasts
314.在天然和重组酵母中鉴定控制从木糖到乙醇的碳通量的新型代谢相互作用
摘要:不同于木糖转化的天然酵母,表达相同木糖同化途径的酿酒酵母的重组菌株在厌氧条件下产生木糖醇而不是从木糖以低特异性木糖转化率产生乙醇。尽管在过去的二十年中进行了大量的研究工作,但这些表型的差异不能用当前的代谢和动力学模型来解释。为了提高我们对木糖碳代谢通量控制乙醇的控制,我们开发了一种基于酶机理的新型动力学模型,并应用定量代谢物分析和酶活性分析一起研究了Candida tenuis CBS4435(木糖代谢的木糖代谢= 0.10g / gdc / h,25℃; Y乙醇= 0.44g / g; Y木糖醇= 0.09g / g)和重组酿酒酵母菌株BP000(q木糖= 0.07g / gdc / h,30℃ ; Y乙醇= 0.24g / g; Y木糖醇= 0.43g / g),全面表达相同的木糖还原酶(XR)。结果:从菌株到菌株代谢对照分析的结果表明XR的活性水平和上部糖酵解(UG;在CBS4435中都≥10倍更高)的最大通量容量主要贡献于表型分化,而来自氧化戊糖磷酸途径扮演次要角色。胞内代谢物谱支持从动力学模型获得的结果,并表明UG代谢物的库大小与通过UG的碳通量之间呈正相关。对于CBS4435,通过UG的快速碳通量可能与果糖-6-磷酸对6-磷酸果糖激酶(PFK)活性的变构控制有关。 CBS4435将UG代谢物保持在高水平的能力可由低甘油3-磷酸磷酸酶(GPP,CBS4435中低17倍)和高XR活性解释。结论:通过应用系统生物学方法,我们将基于动力学建模的代谢控制分析的结果与从定量代谢物分析和酶活性分析获得的数据相结合,我们可以提供有助于控制碳的代谢和动力学相互作用的新见解从木糖到乙醇的通量。在证据的支持下,提出了两个新的目标,即PFK和GPP,除XR之外在表型分化中起关键作用。未来高效快速的微生物乙醇生产者的设计当然可以从这项研究中得到的结果中受益。
315. Inference of Network Dynamics and Metabolic Interactions in the Gut Microbiome
315.肠道微生物组中网络动态和代谢相互作用的推断
摘要:我们提出了一种新的方法,从时间序列宏基因组信息构建一个布尔动态模型,并将此模型与基因组规模的代谢网络重建相结合,以确定微生物相互作用的代谢基础。我们在一个重大的健康问题的背景下应用:克林霉素抗生素治疗和机会性艰难梭菌感染。我们的模型重述克林霉素抗生素治疗和艰难梭菌感染的已知动力学,并预测治疗性益生菌干预以抑制艰难梭菌感染。基因组规模的代谢网络重建揭示了社区成员之间的代谢差异,并用于探索代谢在观察到的微生物相互作用中的作用。体外实验数据验证了我们的计算模型的关键结果,即B. intestinihominis事实上可以减缓艰难梭菌的生长。
316. Inferring microbial interaction network from microbiome data using RMN algorithm
316.利用RMN算法从微生物组数据中推断微生物相互作用网络
摘要:背景:微生物相互作用本质上是无处不在的。最近,已经开发了许多基于相似性的方法来研究微生物生态系统中的相互作用。这些方法只能解释非方向性的相互作用,而更完整地看待微生物如何相互调节仍然是难以捉摸的。结果:本研究采用基于规则的微生物网络(RMN)算法,将规则OTU三重模型与参数加权函数相结合,开发来构建微生物调控网络。 RMN算法不仅可以推断微生物之间的合作关系和竞争关系,而且可以推断出这种相互作用的方向。另外,RMN算法可以从理论上表征微生物网络中微生物对相关系数较低的调控关系。我们的研究结果表明,双歧杆菌,链球菌,梭状芽孢杆菌XI和类杆菌对于引起肠道微生物群中韦荣恩氏菌的丰度变化是必不可少的。此外,我们推测了一些可能的微生物相互作用,包括韦荣拉和拟杆菌的竞争关系,以及韦荣拉和梭菌XI的合作关系。结论:RMN算法提供了肠道微生物网络的重建,可以揭示微生物在婴儿肠道内的动力学相互作用。
317. Inferring microbial interaction networks from metagenomic data using SgLV-EKF algorithm
317.使用SgLV-EKF算法从宏基因组数据中推断微生物相互作用网络
摘要:背景:推断微生物相互作用网络(MINs)并模拟其动力学对于理解细菌生态系统的机制和设计抗生素和/或益生菌疗法至关重要。最近,提出了几种使用广义Lotka-Volterra(gLV)模型推断MIN的方法。这些模型的主要缺点包括这些模型只考虑了测量噪声而没有考虑底层动力学的不确定性。此外,推断MIN的特点是监管机制中有限的观察和非线性。因此,需要新的估算技术来解决这些挑战。结果:本文提出了SgLV-EKF:采用扩展卡尔曼滤波(EKF)算法对MIN动态进行建模的随机gLV模型。特别是,SgLV-EKF采用MIN的随机建模,在动力学模型中增加一个噪声项来补偿建模的不确定性。这种随机建模比传统的gLV模型更为真实,它假定MIN动力学完全由gLV方程控制。在指定随机模型结构之后,我们提出EKF来估计MIN。将SgLV-EKF与基于相似度的两种算法进行了比较,一种是基于积分的算法,另一种是基于回归的算法,在两个合成数据集和两个真实数据集上所取得的性能。第一个数据集模拟测量数据中的随机性,而第二个数据集包含基本动态的不确定性。最近有关抗生素介导的艰难梭菌感染的研究提供了真实的数据集。实验结果表明,SgLV-EKF在测量噪声的鲁棒性,建模误差以及追踪MIN的动力学方面优于其他方法。结论:性能分析表明,所提出的SgLV-EKF算法是一个强大而可靠的工具来推断MINs并跟踪他们的动态。
318. Interaction networks for identifying coupled molecular processes in microbial communities
318.微生物群落中识别偶联分子过程的相互作用网络
摘要:背景:微生物群落适应环境条件以优化代谢通量。这种适应可能包括合作机制,最终导致表型可观察到的作为不能单独归因于个体物种的突然性质。了解跨物种合作的分子基础增加了工业应用中微生物群落的利用,包括金属生物浸出和生物修复过程。随着宏基因组学的显着进步,微生物群落的组成变得易于综合分析涉及多个物种的纠缠分子过程的水平,进而提供了用于分析合作现象的分子基础的数据矩阵。方法:我们提出了一个分析框架与动力学层次概念对齐,以揭示微生物群落中的突发性质,并举例说明了这种方法在At的共同培养环境中的应用。氧化亚铁和At。杆菌。与单个物种的活性相比,这种最小微生物群体表现出生物浸出效率的显着增加,涉及硫代硫酸盐,多硫化物和铁氧化途径的机制。结果:填充具有丰富的功能注释和相互作用信息的以基因为中心的数据结构允许在耦合微生物物种的能量生产和运输过程的功能层面推导网络模型。在包括能量生产和运输的直系同源基因的相互作用网络上应用网络分割方法,提出了一系列与生物浸出相关的特定分子过程。由此产生的分子过程模型主要涉及铁氧化,氮代谢和质子转运等功能,辅之以硫氧化和氮代谢,以及一组离子转运功能。在分子模型表示中嵌入的氧化铁特定基因具有支持氨利用以及生物膜形成的基因功能,类似于在共培养情况下有效黄铜矿生物浸出的关键元素作为应急性质。结论:在分段的,以基因为中心的相互作用网络的水平上分析微生物群落的纠缠分子过程允许识别核心分子过程和功能,这增加了我们对微生物聚生体突变特性的机理理解。
319. Large-scale in silico modeling of metabolic interactions between cell types in the human brain
319.人脑中细胞类型之间的代谢相互作用的大规模计算机建模
摘要:提出了一个整合了基因表达数据,蛋白质组学数据和基于文献的手工定制的工作流程,以构建人脑能量代谢的多细胞,组织特异性模型,重述星形胶质细胞和各种神经元类型之间的代谢相互作用。对基因鉴定,组学数据分析和生理状态分析应用三种分析。首先,我们将谷氨酸脱羧酶鉴定为可能有助于阿尔茨海默病的细胞类型和区域特异性的靶标。其次,在阿尔茨海默氏病的受影响的脑区中观察到的代谢率下降与在组织病理学正常神经元中抑制中央代谢基因表达一致。第三,我们确定胆碱能神经元中的通路,它们将线粒体代谢和细胞溶质乙酰胆碱的产生耦合起来,随后发现胆碱能神经传递占脑神经传递的〜3%。因此,基于约束的建模可以有助于研究和分析人体组织中的多细胞代谢过程,并提供对高通量数据分析的详细的机理洞察。
320. Limitations of a metabolic network-based reverse ecology method for inferring host–pathogen interactions
320.基于代谢网络的逆向生态学方法推断宿主-病原体相互作用的局限
摘要:背景:宿主 - 病原体相互作用在广泛的研究领域中是重要的。鉴于宿主与病原体之间代谢串扰的重要性,提出了基于代谢网络的逆向生态学方法来推断这些相互作用。然而,由于所提出的各种解释和迄今为止被忽略的潜在混杂因素的影响,该方法的有效性仍不清楚。结果:我们重新评估了逆向生态学方法评估宿主 - 病原体相互作用的重要性,同时用氧需求,基因组,代谢网络和系统发育数据统计控制混杂效应。我们的数据分析显示宿主 - 病原体相互作用受基因组大小,主要网络参数(例如边缘数量),需氧量和系统发育的影响比基于储备生态学的措施更强烈。结论:这些结果表明逆向生态学方法的局限性;然而,他们并没有对完全采用逆向生态学方法的重要性进行折扣。相反,我们强调需要开发更合适的推断宿主 - 病原体相互作用的方法,并对代谢网络与宿主 - 病原体相互作用之间的关系进行更仔细的检查。
321. Metabolic Interaction of Helicobacter pylori Infection and Gut Microbiota
321.幽门螺杆菌感染和肠道微生物的代谢相互作用
摘要:作为屏障,消化道共生菌群可以预防胃肠道中潜在的致病微生物。肠道微生物和免疫细胞之间的串扰促进人体肠道动态平衡。2型糖尿病(T2D)中许多人类代谢紊乱如肥胖,肝脂肪肝和胰岛素抵抗的发展与肠道微生物群的肠道菌群失调有关。某些微生物,如丁酸生产细菌,在T2D患者中较低。精益捐赠者肠道微生物群的转移会增加代谢综合征患者的胰岛素敏感性,但确切的发病机制尚不清楚。幽门螺旋杆菌在人的胃中引起慢性胃炎,消化性溃疡和胃癌。幽门螺杆菌感染也诱导胰岛素抵抗,并已被定义为T2D发展的诱发因素。幽门螺杆菌感染者和小鼠的胃和粪便微生物群可能已经改变,以促进胃炎和特定疾病。然而,幽门螺旋杆菌和肠道微生物群在调节宿主代谢中的相互作用也是未知的。进一步的研究旨在确定幽门螺杆菌 - 微生物群 - 宿主代谢轴,并测试幽门螺杆菌根除或肠道微生物群的修饰是否可以改善人类代谢紊乱的控制。
322. Metabolic interactions in microbial communities: untangling the Gordian knot
323.微生物群落中的代谢相互作用:解开Gordian结
摘要:代谢交换在微生物群落中无处不在。然而,由于其内在的动态性和社区的复杂性,检测代谢物交叉饲养是困难的。因此,虽然在自然系统中运作的代谢网络的详尽描述是未来的一项任务,但今天的争斗被分为小而复杂的微生物联合体的详细表征,并集中在自然生态系统的特定代谢方面。检测代谢相互作用需要方法学的混合能够捕捉物种的身份,依赖性和交换代谢物的性质。从宏基因组学到成像质谱技术的多种技术的多种组合,为这一挑战提供了解决方案,每种组合都针对当前社区量身定制。
323. Metabolic network analysis reveals microbial community interactions in anammox granules
323.代谢网络分析揭示厌氧氨氧化颗粒污泥中微生物群落的相互作用
摘要:介导厌氧氨氧化(厌氧氨氧化)的微生物群落是从废水中去除氮气的最节能的环境生物技术之一。然而,关于异养细菌在厌氧氨氧化菌颗粒中起作用的功能知之甚少。在这里,我们使用基因组中心宏基因组学来从实验室规模的厌氧氨氧化生物反应器中恢复17种厌氧氨氧化和异养细菌的基因组草图。我们结合代谢网络重建与宏转录组学审查厌氧氨氧化和异养细菌的基因表达,并确定其潜在的相互作用。我们发现Chlorobi附属细菌可能是高度活跃的蛋白质降解物,分解代谢胞外肽,同时将硝酸盐回收成亚硝酸盐。其他异养生物也可能有助于清除由厌氧氨氧化细菌产生的碎屑和肽,并可能使用替代的电子供体,例如H 2,乙酸盐和甲酸盐。我们的研究结果提高了对厌氧氨氧化菌和异养细菌的代谢活动和相互作用的理解,并提供了关于厌氧氨氧化菌颗粒的生态系统功能的第一个转录见解。
324. MetaMIS: a metagenomic microbial interaction simulator based on microbial community profiles
324.MetaMIS:一个基于微生物群落谱的宏基因组微生物相互作用模拟器
摘要:背景:微生物群落的复杂性和动态是系统生态的主要因素。通过NGS技术,宏基因组学数据为探索微生物相互作用提供了新的途径。已经被广泛用于推断动态系统中动物相互作用的Lotka-Volterra模型最近已被应用于宏基因组数据的分析。结果:本文介绍了基于Lotka-Volterra模型的工具Metagenomic Microbial Interacticon Simulator(MetaMIS),用于分析微生物群落剖面的时间序列数据。 MetaMIS首先从丰富的表格中为操作分类单元(OTU)推断潜在的微生物相互作用,然后使用Lotka-Volterra模型解释相互作用网络。我们还在MetaMIS中嵌入了一个Bray-Curtis相异性方法来评估与生物现实的相似性。MetaMIS旨在容忍高水平的缺失数据,并且可以估计交互信息而不受稀有微生物的影响。对于每个互动网络,MetaMIS系统地检查互动模式(如共生或竞争),并改善微生物中的生物作用。作为一个案例研究,我们收集了一个人类男性粪便微生物组,并且显示相对低丰度OTU的微球菌(Micrococcaceae)与13个主要的OTU高度相关,似乎起到关键作用。MetaMIS能够将多个交互网络组织成一个共识网络进行比较研究;因此我们作为案例研究也已经确定了雌性和雄性粪便微生物群之间的共识相互作用网络。结论:MetaMIS提供了一个高效和用户友好的平台,可以揭示宏基因组学数据的新见解。MetaMIS免费提供:https://sourceforge.net/projects/metamis/。
325. Microbial interactions for enhancement of a-amylase production by Bacillus amyloliquefaciens 04BBA15 and Lactobacillus fermentum 04BBA19
325.用于增强由解淀粉芽孢杆菌04BBA15和发酵乳杆菌04BBA19产生α-淀粉酶的微生物相互作用
摘要:将酿酒酵母和两种耐热α-淀粉酶生产菌株(解淀粉芽孢杆菌04BBA15和发酵乳杆菌04BBA19)之间发生的相互作用通过比较它们在分离中获得的生长模式与在混合物中获得的生长模式进行分析。使用方差分析(ANOVA)评估模式之间的差异,以测量生物体的生长受其他因素影响的程度。结果显示混合培养中有两种类型的相互作用;酿酒酵母和解淀粉芽孢杆菌04BBA15之间的共生作用以及酿酒酵母和发酵乳杆菌04BBA19之间的共生作用。在混合培养中,α-淀粉酶的产量比单种培养显着增加(P <0.05)。混合培养物中发酵参数的响应表面优化(初始酵母与细菌比例1.125,温度33.5℃,pH 5.5)导致比未优化的发酵中观察到的高约1.8倍的酶产量。
326. Microbial interactions: from networks to models
326.微生物相互作用:从网络到模型
摘要:宏基因组学和16S焦磷酸测序技术使得生态系统结构和动力学的研究有了很大的深度和准确性。在这些数据集中发现的共现和相关模式越来越多地用于预测从海洋到人类微生物组的环境中的物种相互作用。此外,平行共培养分析和组合标记实验允许高通量发现物种间的合作和竞争关系。在这篇综述中,我们描述了这些技术如何为全球生态系统网络预测和全生态系统动态模型的发展开辟道路。
327. MIMO: an efficient tool for molecular interaction maps overlap
327.MIMO:一个用于分子交互图重叠的有效工具
摘要:背景:分子通路代表细胞内和细胞之间的分子间发生的相互作用的整体。跨越生物体和功能的分子途径之间的相似性的鉴定对理解复杂的生物学过程具有关键作用。为了推断这种新颖的信息,分子途径的比较需要考虑不完美的匹配(灵活性)并且有效地处理复杂的网络拓扑。迄今为止,这些特征仅在部分用于比较分子相互作用图谱的工具中可用。结果:我们的方法MIMO(分子相互作用图重叠)解决了第一个问题,允许在查询和模板路径之间引入空位和不匹配,并且允许在需要监督的查询中结合先验生物信息。然后直接依靠系统生物学标记语言(SBML)标准中描述的丰富的图形拓扑来解决第二个问题,并使用多重图形高效地处理生物图数据库上的多个查询。这个算法已经成功地用于突出Reactome数据库中各个人类途径之间的接触点。结论:MIMO为比较复杂的生物通路提供了灵活而高效的图形匹配工具。
328. MMinte: an application for predicting metabolic interactions among the microbial species in a community
328.MMinte:一个预测群落中微生物物种之间代谢相互作用的应用
摘要:背景:微生物研究的爆炸式增长已经产生了大量的数据。这些数据为我们提供了许多答案,但也提出了许多问题。16S rDNA-微生物组分析的骨干-使我们能够评估表征生态系统整体性质的α-多样性,β-多样性和微生物 - 微生物关联。然而,我们仍然无法使用16S rDNA数据直接评估确定该系统更广泛生态的微生物 - 微生物和微生物 - 环境相互作用。因此,诸如竞争,合作和营养条件等性质仍然没有充分分析。在这里,我们应用16S rDNA数据确定的微生物预测社区代谢模型来探索微生物群落的生态。结果:我们开发了一个从16S rDNA数据大规模评估微生物代谢相互作用(MMinte)的方法。MMinte评估群落代谢网络中相互作用的生物对的相对增长率,以及这种相互作用是否有积极或消极的影响。此外,MMinte的模拟考虑了营养环境,在确定个体微生物的代谢方面起着重要的作用。我们提出了两个案例研究来证明这个软件的实用性。首先,我们展示饮食如何影响微生物 - 微生物相互作用的性质。第二,我们使用MMinte的模块化功能集,以更好地了解如何在代谢条件下,Desulfovibrio piger的增长受简化肠道社区中其他成员的影响,并影响其他成员的增长,这些因素被认为是其动态的决定因素。结论:通过将代谢模型应用于常用的序列数据,MMinte授权用户洞察微生物之间的代谢关系,强调可能与生态稳定性,易感性和交叉喂养有关的重要特征。这些关系是影响我们理解诸如微生物衍生的结肠癌毒性等问题的能力的广泛生态问题的基础。
48. Model-based quantification of metabolic interactions from dynamic microbial-community data
48.动态微生物群落数据的代谢相互作用的基于模型的量化
摘要:微生物生态学面临的一个重要挑战是从群落级数据,关于物种丰度和代谢物浓度推断生长的微生物物种之间的代谢交换通量。在这里,我们应用基于模型的方法来整合这些实验数据,从而推断代谢交换通量。我们设计了丙酮丁醇梭菌和Wolinella succinogenes的合成厌氧共培养物,它们通过种间氢转移相互作用并应用不同的环境条件,我们预期代谢交换速率会改变。我们使用代表我们目前的生理学理解的两种微生物代谢模型,发现这种理解 - 模型 - 足以推断代谢交换通量的身份和大小,并且它意味着意想不到的相互作用。如果模型不适合所有的实验数据,则表示进一步的生理学研究的具体要求。我们表明氮源影响共同培养物种间氢转移的速率。此外,该模型可以预测细胞内通量和最佳代谢交换率,这可以指向工程策略。因此,这项研究提供了一个现实的说明,基于模型的异质数据整合的优势和弱点,从群落水平的实验数据推断代谢交换通量的可能性。
76. Multispecies communities: interspecies interactions influence growth on saliva as sole nutritional source.
76.多物种群落:种间相互作用影响唾液作为唯一营养来源的生长
摘要:人类口腔细菌生活在被称为牙菌斑的生物膜的多个物种群落中。这篇综述重点讨论了七种物种的相互作用以及每种物种单独和与其他物种一起在唾液上生长作为唯一营养来源的能力。 使用物种特异性荧光团缀合的免疫球蛋白G来监测流动细胞中生物膜的群落形成,并且通过共焦显微镜来捕获图像。早期殖民veillonellae出现在唾液的种间相互作用的审查作为一个重要的属,指导多物种群落的发展。高度选择性的种间识别是显而易见的,因为最初的定殖者与早期和中期的定居者配对以形成在唾液上生长的多种群。
329. PID: the Pathway Interaction Database
329.PID:通路相互作用数据库
摘要:Pathway Interaction Database(PID,http://pid.nci.nih.gov)是由人类分子信号和调控事件以及关键的细胞过程组成的可自由获取的策划和同行评审的途径。该数据库由美国国家癌症研究所和自然出版集团合作创建,作为癌症研究团体和其他对细胞通路感兴趣的研究工具,如神经科学家,发育生物学家和免疫学家。PID提供了一系列的搜索功能来促进路径探索。用户可以浏览预定义的一组路径,或创建以感兴趣的单个分子或细胞过程为中心的交互网络图。此外,批量查询工具允许用户上传分子的长列表,例如来自微阵列实验的分子,并将这些分子覆盖到预定义的路径上或可视化完整的分子连接图。用户还可以用可扩展标记语言(XML)和生物通路交换(BioPAX)2级格式下载分子列表,引用列表和完整的数据库内容。数据库每月更新一次新的途径内容,并辅之以其他相关在线工具实际使用的专门委托文章。
16. Predicting microbial interactions through computational approaches
16.通过计算方法预测微生物相互作用
摘要:微生物在各种生态系统中起着至关重要的作用,它们之间的相互作用是理解微生物群落组织和功能的重要一步。计算预测最近已经成为调查微生物相互作用的广泛使用的方法。我们对由他们使用的数据类型组织的新兴计算方法进行了全面的回顾。我们强调使用宏基因组调查数据推断相互作用的三个主要挑战,并讨论相互作用推理算法的基本假设和数学。另外,我们回顾了依赖于代谢途径的交互预测方法,这些方法越来越多地用于揭示相互作用的机制。此外,我们还强调挖掘科学文献对于微生物相互作用的重要性 - 这是一个被大量忽视的实验验证相互作用的数据源。
330. The integrated analysis of metabolic and protein interaction networks reveals novel molecular organizing principles
330.代谢和蛋白质相互作用网络的综合分析揭示新的分子组织原则
摘要:背景:生物相互作用网络的研究是系统生物学的中心议题。在这里,我们调查两种不同类型的相互作用网络之间的关系:代谢途径图和蛋白质 - 蛋白质相互作用网络(PIN)。人们早已确定,连续的酶促步骤通常由形成永久性或瞬时多酶复合物的物理相互作用的蛋白质催化。检查高通量PIN数据,最近显示,事实上,参与连续反应的酶通常比其他蛋白质对更可能相互作用。在我们的研究中,我们扩展了这一研究领域,以包括各自的网络拓扑结构的比较,以及调查酶相互作用的空间组织是否与代谢效率相关。结果:分析酵母数据,我们检测到两种网络类型中蛋白质之间的最短路径之间的长距离相关性,表明两种网络架构相互对应。我们发现代谢酶之间物理相互作用的组织原理与所有蛋白质的一般PIN不同。尽管蛋白质之间的物理相互作用通常是不可逆的,但是观察到酶相互作用是相辅相成的。因此,酶经常与其他相似但不同程度的酶相互作用。携带高通量负荷的酶比代谢通量较低的酶更可能与物理相互作用。具体而言,发现与分解代谢途径有关的酶以及参与复合物分子生物合成的酶显示高度的物理聚集。鉴定了连接细胞代谢的主要组分的单一蛋白质,因此可能对于几个生物合成系统的结构完整性是必不可少的。结论:我们的结果揭示了蛋白质相互作用网络和代谢途径网络之间的拓扑等价性。进化蛋白质相互作用可能通过允许更高的代谢通量显着地提高代谢过程的效率。因此,我们的结果进一步阐明了塑造功能(代谢)以及物理交互网络的统一原则。
14. The large-scale organization of the bacterial network of ecological co-occurrence interactions
14.生态共现相互作用细菌网络的大规模组织
摘要:在自然环境中,微生物形成复杂的相互作用系统。如果忽视细菌群落的结构和组织,可能会带来广泛的医疗和生态后果,但目前还缺乏对环境相互作用网络的全面描述。在这里,我们利用科学文献中的同现来构建这样一个网络,并且展示了物种生活方式与共同发生伙伴的记录数量之间的预期联系模式。我们进一步关注注释良好的肠道群落,并显示典型肠道细菌的大多数同现交互作用发生在该群落内。然后将网络聚类成与自然发生的群落显着对应的物种群。群体内资源竞争,代谢产量和增长率之间的关系符合r / K选择理论。总的来说,这些结果支持构建的群集作为细菌生态系统模型的第一个近似值。预测群落的全面收集形成了一个新的数据资源,进一步系统地刻画群落的生态设计原则。在这里,我们展示了它用于预测群落内的合作和抑制的效用。
331. The linear neighborhood propagation method for predicting long non-coding RNA –protein interactions
331.用于预测非编码RNA-蛋白质相互作用的线性邻域传播方法
摘要:长的非编码RNA(lncRNA)因其在各种生物过程中的基本功能而受到广泛的关注。尽管大多数lncRNA的精确功能和机制仍然未知,但是研究显示lncRNA通常通过与相应的RNA结合蛋白相互作用而发挥功能。lncRNA-蛋白质相互作用的实验检测是昂贵和耗时的。在本文中,我们提出一种线性邻域传播方法(LPLNP)来预测lncRNA-蛋白质相互作用。 LPLNP计算特征空间中的线性邻域相似度,并将其转移到交互空间中,并通过标签传播过程预测lncRNA与蛋白质之间的不可观测的相互作用。我们的研究表明,基于已知的lncRNA-蛋白质相互作用的LPLNP模型可以产生高精度的表现,达到0.42的AUPR得分。此外,我们将lncRNAs和蛋白质的生物信息纳入到LPLNP模型中,并且可以进一步提高性能,达到0.4584的AUPR得分。该案例研究表明,我们的方法预测的许多lncRNA-蛋白质相互作用可以验证,表明我们的方法是lncRNA-蛋白质相互作用预测的一个有用的工具。本文中使用的源代码和数据集可在以下网址获得:https://github.com/BioMedicalBigDataMiningLabWhu/lncRNA-protein-interaction-prediction。
332. Understanding the interactions between bacteria in the human gut through metabolic modeling
332.通过代谢建模了解人类肠道细菌之间的相互作用
摘要:人类肠道微生物组在维护人类健康方面起着举足轻重的作用,是疾病防治的潜在目标。基因组规模的代谢模型(GEMs)可以增加对饮食影响背后的机制,基因型 - 表型关系和微生物稳健性的理解。在这里,我们重建了三个关键物种(Bacteroides thetaiotamicron,Eubacterium rectale和Methanobrevibacter smithii)作为三个主要门在人类肠道(Bacteroidetes,Firmicutes和Euryarchaeota)的相关代表GEMs。我们模拟了这三种细菌在不同肠道生态系统组合中的相互作用,并将预测结果与无菌小鼠定植得到的实验结果进行了比较。此外,我们使用我们的GEM来分析每个物种对基于转录组数据的肠道微生物群整体代谢的贡献,并证明这些模型可以用作理解肠道中细菌相互作用的支架。
333. Visualization of Metabolic Interaction Networks in Microbial Communities Using VisANT 5.0
333.VisANT5.0可视化微生物群落中的代谢相互作用网络
摘要:微生物群落中的代谢网络的复杂性提出了一个未解决的可视化和解释挑战。我们在用于分析生物网络的软件工具VisANT 5.0的新扩展版本中解决了这个挑战。我们特别关注促进社区中微生物之间代谢相互作用的视觉探索,例如,正如COMETS(Computation of Microbial Ecosystems in Time and Space)所预测的,一个动态的化学计量模型框架。使用VisANT独特的metagraph实现,我们展示了如何使用VisANT 5.0来探索不同的时间依赖的生态系统级代谢网络。具体来说,我们分析了以前显示的两种细菌之间的代谢相互作用网络,以显示专性交叉互作的依赖性。此外,我们说明了一个假定的最小的肠道微生物群落如何可以在我们的框架中代表,使突出的多个共存物种的相互作用成为可能。我们设想,VisANT的“共生布局”可以作为复杂微生物群落以及异质人体组织代谢分析的一般工具。 VisANT可在http://visant.bu.edu和COMETS http://comets.bu.edu免费获得。
364. Interaction networks, ecological stability, and collective antibiotic tolerance in polymicrobial infections
364.相互作用网络、生态稳定性以及多菌感染中的集体抗生素耐受性
摘要:多种微生物感染构成了适应几种细菌种类的小型生态系统。通常,这些细菌被孤立地研究。然而,在存在和不存在抗生素的情况下,菌株之间相互作用的程度以及它们之间的相互作用是否会改变细菌的生长和生态系统的抗性还不清楚。我们量化了从23个被诊断为多细菌尿路感染的个体收集的72个细菌分离株的完整生态相互作用网络,发现大多数相互作用基于进化相关性聚类。统计网络分析表明,全球网络中富有竞争性和合作性的互惠互动,而个体寄宿社区网络中的合作互动则被耗尽。我们的测量参数化的种群动态模型表明,相互作用限制了群落的稳定性,解释了观察到的这些群落的物种多样性。我们进一步表明,临床分离物经常相互保护临床相关的抗生素。总之,这些结果强调,生态相互作用对细菌在多种微生物感染群体中的生长和存活至关重要,并影响其组装和恢复能力。
366. Lotka-Volterra pairwise modeling fails to capture diverse pairwise microbial interactions
366.Lotka-Volterra成对建模不能捕获不同的成对微生物相互作用
摘要:成对模型通常用来描述多种群落。在这些模型中,个体通过与社区中每个物种的成对相互作用(“可加性假设”)接受加性适应效应。所有的成对相互作用通常用一个方程来表示,其中参数反映适应效应的标志和强度(“普遍性假设”)。在这里,我们显示一个单一的方程不能定性地捕获不同的配对微生物相互作用。我们为参与常见化学介导的相互作用的两种微生物物种建立机械参考模型,并试图推导出成对模型。不同的方程式取决于介体是可消耗还是可重复使用,相互作用是否由一个或多个介体介导,有时甚至是社区的定量细节(例如,两种物种的相对适合度,初始条件)是适当的。我们的研究结果,加上在许多物种群体中可能违反可加性假设,表明配对建模常常不能预测微生物动态。